こんにちは! Strategic AI Groupの金子です。
夏の自由研究ブログ連載2021として医薬品副作用データベースにWord2Vecを適用し性能を評価、医薬品-原疾患-有害事象の可視化を行いました。
はじめに
本記事で作成したプログラムは医薬品の情報を扱っていますが、医療機器には該当せず人の疾病の診断、治療、予防を目的としておりません。本記事の内容に基づいて医学的判断は行わないようお願いします。
この記事は「用語解説」・「実装」・「実験と評価」・「Embedding projectorによる可視化」の4章に分かれております。
- 「用語解説」の章ではこの記事を書いた背景と記事で出てくる用語について解説しています。
- 「実装」の章では実装に用いたデータ・手法とそのコードについて記載しています。
- 「実験と評価」の章では実験の定量評価と、得られた医薬品同士の結果について考察を行っています。
- 「Embedding projectorによる可視化」の章ではウェブアプリを用いた可視化の方法について紹介しています。
サムネイルの結果を確認したい場合はPublish済みのEmbedding Projectorのページからすぐに見ることができます。
背景・用語解説
背景・概要について
今回の自由研究は、Word2Vecを自然言語以外の分野に適用する際の改善手法の検討と、結果の可視化方法の勉強を目的として行いました。
Word2Vecは自然言語処理以外にも使われており、ECサイトの閲覧履歴を文章として扱うことで学習し獲得した商品の分散表現を協調フィルタリングに活用するItem2Vecのような手法が研究されています。それらの手法では推薦精度をもってモデルの評価を行いますが、得られた分散表現の自体の類似度の評価についてではないためその分散表現を用いてデータの分析を行えるかは分からないといった問題があります。本記事では薬効分類番号を用い、医薬品の分散表現の評価を行うことでWord2Vecに対する様々な改善が分析の役に立つかについて定量的に評価を行いました。また、医薬品と原疾患などの関係は医薬品の添付文章などを参照することで検討できるため、定量評価の妥当性についても考察しました。
用語解説
Word2Vecとは
Word2Vecは自然言語処理でよく使われる、単語を表現する固定長のベクトルを学習するための手法です。ある単語とその周囲の単語の関係からベクトルを学習することで、例えば下記の図のように国と首都の関係性などを表現できるようになります。1
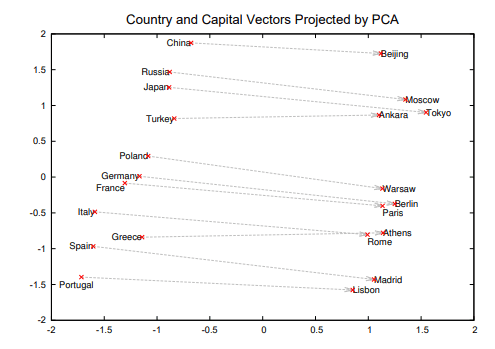
また、Word2Vecで得られた表現には加法構築性があります。学習した単語の分散表現同士で足し算する例(「王様」-「男」+「女」 = 「女王」)は聞いたことがある人も多いと思います。この性質から、前述のとおり自然言語以外の分野でもWord2Vecは広く使われています。
医薬品副作用データベース JADERとは
医薬品副作用データベース(Japanese Adverse Drug Event Report database:JADER)は独立行政法人 医薬品医療機器総合機構(以下PMDA)が公表している「副作用が疑われる症例報告に関する情報」についてのデータベースです。このデータセットは平成25年度以降に医療機関等から報告のあった国内の副反応疑い報告が調査・研究のために利用できるようcsv形式によりまとめられています。
1つの症例報告は以下の4つのテーブルに分割され、それぞれ識別番号によって紐づけられます。
- 患者の年齢や性別、報告者の資格といった症例一覧テーブル(demoテーブル)
- 症例発症時に同時に併用していた薬を含む医薬品情報テーブル(drugテーブル)
- 発生した副作用が疑われる有害事象についての副作用テーブル(reacテーブル)
- 患者がもともと持っていた疾患に関する原疾患テーブル(histテーブル)
今回は、2021/7/23にPMDAの医薬品副作用データベース(英名:Japanese Adverse Drug Event Report database、略称;JADER)から取得したデータを用い実験を行いました。
1つの識別番号によって結びついた情報を1つの文章として捉えWord2Vecを実装します。
薬価基準収載医薬品コードとは
薬価基準収載医薬品コードは厚生労働省が管理する薬価ごとに設定された英数12桁のコードです。薬価基準収載医薬品コードのうち上位4桁は薬効分類番号で日本標準商品分類の中分類87の次の4桁に対応しています。例えばアスピリンには1143と3399が振られており、これは以下のような意味を持ちます。分類検索システムで実際に検索できます。
1143: 神経系及び感覚器官用医薬品 > 中枢神経系用薬 > 解熱鎮痛消炎剤 > サリチル酸系製剤;アスピリン等 |
JADER記載の医薬品名と薬価基準収載医薬品コードを紐づけるために厚生労働省のHPからデータを取得しました。
薬価基準収載医薬品コードの出典は2021/8/17に取得した以下のページです。
また、薬効分類の名称については総務省のHPからデータを取得しました。
日本標準商品分類の出典は2021/8/17に取得した以下のページです。
これらのページに記載されたファイルについて編集・加工を行い計算に用いました。
実装
この章ではデータセットの基本的な統計情報と実装した手法の解説、コードの紹介を行います。
データセット基本情報
JADERのデータセットについて、693295件の報告を元に学習を行いました。
各テーブルに含まれるユニークな語彙数とレコード数は以下の通りです。
| ユニークな語彙数 | レコード数 | |
|---|---|---|
| 医薬品情報テーブル | 10106 | 3875874 |
| 副作用テーブル | 9681 | 1096193 |
| 原疾患テーブル | 9903 | 1392614 |
医薬品の名称のうち、2071件は薬効分類番号との紐づけができたため、この2071件の類似度を用いて得られたベクトルの評価を行います。紐づけができた薬効分類番号は377種類ありました。
手法解説
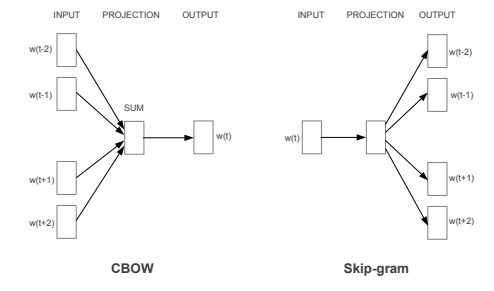
Skip-gramとCBOW
Word2Vecの学習で現在よく使われる手法は二種類あります。周囲の単語からある単語を予測するCBOW(Countinuous Bag-of-Words)モデルと、ある単語から周囲の単語を予測するSkip-gramモデルです。一般にCBOWよりもSkip-gramの方が計算時間はかかる分精度が高いのですが、実装が簡単であるのと拡張しやすいことから本記事ではCBOWをベースに実装を行います。2
Negative Sampling
CBOWもSkip-gramも多クラス分類としてN種類の単語のどれに分類されるか毎回計算することは可能です。しかし、今回の場合単語の種類が30000ほどあるので毎回30000種類の分類の確率を計算するのでは時間がかかってしまいます。そこでサンプルごとにすべての負例を計算するのではなく、ある分布からランダムに5~20程度負例をサンプリングして計算を行うことで高速化します。負例は以下の分布からサンプリングします。
alphaはgensimのデフォルトパラメータだと0.75です。αは1だと元の分布そのままで、小さくなるほどレアなサンプルを重視するようになります。今回はalpha=0.75で統一しました。
nDCG
今回の評価指標はnormalized Discounted Cumulative Gain(nDCG)によって行います。nDCGは検索やレコメンド等でよく使われるランキング手法で、理想的な順位付けにどれくらい近い結果が出せたかをランキングの下位ほど重みを小さくしながら計算します。
具体的な計算式は以下の通りです。
relは関連度で、今回は薬効分類番号が何段階一致しているかで計算します。relの最大はすべて一致している時で2^4-1=15、最小は全て一致していない時で0です。DCGはすべての組み合わせで計算することは計算時間的に困難なため、ランキングの上位K個で評価します。今回はK=100のnDCG@100で計算しました。
コード
ライブラリの読み込みとデータセットの読み込み
import os |
データセットの用意
今回はgensimによる実装とtensorflowによる実装を行います。gensimは各単語を文字列で、tensorflowではidに変換した上で用意する必要があります。ただしgensimは自然言語処理用に作られているライブラリなので、今回のように語順の関係ないデータを食わせる場合は事前にshuffleするといった工夫が必要です。
# 一つの症例に対し重複した有害事象を除く |
評価指標の用意
nDCGの評価の為にデータを加工します。
import re |
メタデータの用意
import csv |
tensorflow用モジュールの実装
Samplerの実装
Tensorflowの学習用のデータを作成するsamplerを実装します。正例は以下の様に実装します。
class OneVsOtherSampler(): |
negative sampler
負例は与えられた分布を元に二分探索を用いながらサンプリングします。
from random import sample |
model
embeddingの抽出部分とCBOWの部分を分離します。
class EmbeddingModel(tf.keras.Model): |
gensim用学習コード
from gensim.models import word2vec |
tensorflow用学習用コード
from adabelief_tf import AdaBeliefOptimizer |
実験と評価
学習と定量評価
実験1: 医薬品の情報のみで学習 vs 医薬品+副作用+原疾患で学習
まずはgensimを用いてデータセットに関する検討を行います。
word2vecについて医薬品の情報のみで学習させたものと副作用、原疾患の情報を加えて学習させたもので比較を行いました。
学習のパラメータについては以下のように設定し、後はgensimのデフォルトパラメータを用いました。
| パラメータ名 | 値 | |
|---|---|---|
| 0 | epoch | 10 |
| 1 | vector_size | 128 |
| 2 | min_count | 1 |
| 3 | window | 15 |
| 4 | workers | 6 |
| 5 | negative | 15 |
医薬品のベクトルについてコサイン類似度を用い近傍を取得し評価した結果、nDCG@10はそれぞれ以下のようになりました。
| モデル名 | nDCG@100 | |
|---|---|---|
| 0 | gensim 医薬品のみ | 0.3883 |
| 1 | gensim 副作用+原疾患あり | 0.4030 |
医薬品の類似度を計算する場合でも、医薬品以外の情報も合わせて学習を行うことで精度が上がることが分かりました。
実験2: 医薬品+副作用+原疾患のデータについてTensorFlowで実装
上記の実験を元に、三種類のデータを用いてtensorflowで学習させたものと比較しました。
ただし、tensorflowでの学習はepoch数を2にし、batch sizeは8192にした上で、後のパラメータはgensimに揃えました。
結果は
| モデル名 | nDCG@100 | |
|---|---|---|
| 2 | tensorflow 副作用+原疾患あり | 0.4191 |
となり、TensorFlow実装の方が良いスコアとなりました。
実験3: Tensorflowモデルのチューニング
tensorflowモデルに以下の改善を行いました。
- Sharpness-Aware Minimization及びAdaptive Sharpness-Aware Minimizationの実装
- バッチサイズを8192 -> 65536に変更(Gradient Accumulationを使用)
- エポック数を2 -> 20に変更
- Warmup + Cosine Annealingによる学習率管理
- Metric learningの追加(AdaCosベース独自実装)
| モデル名 | nDCG@100 | |
|---|---|---|
| 3 | nadare baseline | 0.4640 |
自然言語以外に適用するWord2Vecにおいても、一般的な深層学習のテクニックが有効であることが分かりました。
実験結果まとめ
全ての結果をまとめるとこのようになります。
| モデル名 | nDCG@100 | |
|---|---|---|
| 0 | gensim 医薬品のみ | 0.3883 |
| 1 | gensim 副作用+原疾患あり | 0.4030 |
| 2 | tensorflow 副作用+原疾患あり | 0.4191 |
| 3 | nadare baseline | 0.4640 |
これらの結果のうち、1番と3番についてembeddingの評価を行っていきます。
類似ベクトルの確認と考察
出現数の多い医薬品のなかから薬効分類番号が1つのみ振られているものについて、薬効分類番号の上一桁が被らないよう出現数順に選び、コサイン類似度で近傍を取得しました。選んだ医薬品は以下になります。
| 医薬品(一般名) | 薬効分類番号 | 1 | 2 | 3 | 4 | |
|---|---|---|---|---|---|---|
| 20 | アセトアミノフェン | 1141 | 神経系及び感覚器官用医薬品 | 中枢神経系用薬 | 解熱鎮痛消炎剤 | アニリン系製剤;メフェナム酸,フルフェナム酸等 |
| 2 | アムロジピンベシル酸塩 | 2171 | 個々の器官系用医薬品 | 循環器官用薬 | 血管拡張剤 | 冠血管拡張剤 |
| 27 | アロプリノール | 3943 | 代謝性医薬品 | その他の代謝性医薬品 | 痛風治療剤 | アロプリノール製剤 |
| 26 | フルオロウラシル | 4223 | 組織細胞機能用医薬品 | 腫瘍用薬 | 代謝拮抗剤 | フルオロウラシル系製剤 |
| 855 | 五苓散 | 5200 | 生薬及び漢方処方に基づく医薬品 | 漢方製剤 | ||
| 57 | リバビリン | 6250 | 病原生物に対する医薬品 | 化学療法剤 | 抗ウィルス剤 | |
| 213 | イオパミドール | 7219 | 治療を主目的としない医薬品 | 診断用薬(体外診断用医薬品を除く)。 | X線造影剤 | その他のX線造影剤 |
| 93 | オキシコドン塩酸塩水和物 | 8119 | 麻薬 | アルカロイド系麻薬(天然麻薬) | あへんアルカロイド系麻薬 | その他のあへんアルカロイド系麻薬 |
医薬品同士の類似度の確認
選んだ医薬品と類似した医薬品を探すため、コサイン類似度で近傍を取得しました(【】内は薬効分類番号)
gensim 副作用+原疾患あり
| 0 | 1 | 2 | 3 | 4 | 5 |
|---|---|---|---|---|---|
| アセトアミノフェン【1141】 | ロキソプロフェンナトリウム水和物【1149,2649】 | 炭酸水素ナトリウム・無水リン酸二水素ナトリウム【2359】 | アズレンスルホン酸ナトリウム水和物【2323,2260,2399】 | ペンブロリズマブ【】 | 1141001【】 |
| アムロジピンベシル酸塩【2171】 | ニフェジピン【2171】 | ベニジピン塩酸塩【2171】 | シルニジピン【2149】 | アゼルニジピン【2149】 | エホニジピン塩酸塩エタノール付加物【2149】 |
| アロプリノール【3943】 | フェブキソスタット【3949】 | トピロキソスタット【3949】 | ベンズブロマロン【3949】 | 痛風治療剤【】 | プロベネシド【3942】 |
| フルオロウラシル【4223】 | 5-FU【】 | カペシタビン【4223】 | 1181107【】 | テガフール・ギメラシル・オテラシルカリウム配合剤【4229】 | 5-F【】 |
| 五苓散【5200】 | 桂枝茯苓丸【】 | 当帰芍薬散【】 | 真武湯【】 | 半夏厚朴湯【】 | 柴胡桂枝湯【】 |
| リバビリン【6250】 | レジパスビル アセトン付加物・ソホスブビル【】 | ソラフェニブ【】 | ribavirin【】 | グレカプレビル水和物・ピブレンタスビル【6250】 | prednisolone valerate acetate【】 |
| イオパミドール【7219】 | イオヘキソール【7219】 | イオメプロール【7219】 | イオベルソール【7219】 | イオプロミド【7219】 | ガドブトロール【7290】 |
| オキシコドン塩酸塩水和物【8119】 | モルヒネ硫酸塩水和物【8114】 | トラマドール塩酸塩【1149】 | ヒドロモルフォン塩酸塩【8119】 | フェンタニル【8219】 | モルヒネ塩酸塩水和物【8114】 |
nadare baseline
| 0 | 1 | 2 | 3 | 4 | 5 |
|---|---|---|---|---|---|
| アセトアミノフェン【1141】 | ロキソプロフェンナトリウム水和物【1149,2649】 | UNKNOWNDUG【】 | セフカペン ピボキシル塩酸塩水和物【】 | 麻黄湯【】 | ラニナミビルオクタン酸エステル水和物【6250】 |
| アムロジピンベシル酸塩【2171】 | カンデサルタン シレキセチル【2149】 | バルサルタン【2149】 | オルメサルタン メドキソミル【2149】 | ニフェジピン【2171】 | テルミサルタン【2149】 |
| アロプリノール【3943】 | クエン酸カリウム・クエン酸ナトリウム水和物【3949】 | ベンズブロマロン【3949】 | 球形吸着炭【3929】 | プロベネシド【3942】 | 中枢神経系用薬【】 |
| フルオロウラシル【4223】 | レボホリナートカルシウム【3929】 | オキサリプラチン【4291】 | イリノテカン塩酸塩水和物【4240】 | アフリベルセプト ベータ(遺伝子組換え)【】 | パニツムマブ(遺伝子組換え)【】 |
| 五苓散【5200】 | 真武湯【】 | 柴苓湯【】 | 桂枝茯苓丸【】 | 補中益気湯【】 | 人参養栄湯【】 |
| リバビリン【6250】 | ペグインターフェロン アルファ-2b(遺伝子組換え)【】 | シメプレビルナトリウム【】 | テラプレビル【】 | ペグインターフェロン アルファ-2a(遺伝子組換え)【】 | ソホスブビル【6250】 |
| イオパミドール【7219】 | イオメプロール【7219】 | イオヘキソール【7219】 | イオベルソール【7219】 | イオプロミド【7219】 | アミノ安息香酸エチル・パラブチルアミノ安息香酸ジエチルアミノエチル塩酸塩【2710】 |
| オキシコドン塩酸塩水和物【8119】 | プロクロルペラジンマレイン酸塩【1172】 | モルヒネ硫酸塩水和物【8114】 | フェンタニル【8219】 | モルヒネ塩酸塩水和物【8114】 | ナルデメジントシル酸塩【2359】 |
考察
医薬品間の類似度については、薬効分類番号から分かるように類似した薬物が取れています。薬効分類番号が異なるのに上位に来ている薬物、例えばフルオロウラシルとレボホリナートカルシウムについてですが、レボホリナートカルシウムにはフルオラシルと併用することで抗腫瘍効果を高める効果があるそうです。他にも目視で結果を確認したところ、多剤併用療法が用いられる医薬品については薬効分類番号が近くない場合でも上位に類似した医薬品が現れる傾向が確認できました。
医薬品-原疾患の類似度の確認
医薬品と原疾患のベクトルについてコサイン類似度で近傍を取得しました。
gensim 副作用+原疾患あり
| 0 | 1 | 2 | 3 | 4 | |
|---|---|---|---|---|---|
| アセトアミノフェン | 過敏症 | 脾臓辺縁帯リンパ腫第3期 | 回復期患者 | ドーパ反応性ジストニア | 矮小腎 |
| アムロジピンベシル酸塩 | 視床出血 | 一過性脳虚血発作 | 腫瘍随伴性ネフローゼ症候群 | 脳幹出血 | 拡張期血圧低下 |
| アロプリノール | 尿毒症性アシドーシス | 高血圧 | 酸素消費量 | 高カリウム血症 | 腎硬化症 |
| フルオロウラシル | 子宮頚部癌第4期 | 好中球減少性感染 | 遠隔転移を伴う肝癌 | 腹膜転移 | 遠隔転移を伴う肛門癌 |
| 五苓散 | 補充現象 | 偽アルドステロン症 | 硬膜下血腫除去 | 閉経期症状 | 中毒性皮疹 |
| リバビリン | 肝炎ウィルス関連腎症 | B型肝炎e抗原陰性 | B型肝炎e抗原陽性 | 耳管炎 | 予防 |
| イオパミドール | ヘパリン中和療法 | 過期産児 | 腎病変部切除 | アルコールによる宿酔 | 橈骨動脈脈拍異常 |
| オキシコドン塩酸塩水和物 | 癌疼痛 | 腫瘍熱 | 免疫性腸炎 | 肝転移 | 中枢神経系転移 |
nadare baseline
| 0 | 1 | 2 | 3 | 4 | |
|---|---|---|---|---|---|
| アセトアミノフェン | 発熱 | 口腔咽頭痛 | インフルエンザ | 上咽頭炎 | ウィルス感染 |
| アムロジピンベシル酸塩 | 高血圧 | 高脂血症 | 糖尿病 | 脳出血 | 視床出血 |
| アロプリノール | 高尿酸血症 | 痛風 | 血中尿酸増加 | 痛風性関節炎 | コケーン症候群 |
| フルオロウラシル | 再発直腸癌 | 再発結腸癌 | 食道扁平上皮癌 | 遠隔転移を伴う直腸S状結腸癌 | 肛門扁平上皮癌 |
| 五苓散 | 硬膜下血腫 | 補充現象 | 脛骨内側過労性症候群 | 冷感 | 頭蓋骨骨折 |
| リバビリン | 慢性C型肝炎 | ウィルス血症 | C型肝炎 | 腟膿瘍 | C型肝炎RNA陽性 |
| イオパミドール | コンピュータ断層撮影 | 肝新生物 | 膵新生物 | 大動脈造影 | 尿路造影 |
| オキシコドン塩酸塩水和物 | 癌疼痛 | 骨転移 | 腫瘍熱 | 歯肉損傷 | 骨痛 |
考察
医薬品-原疾患との関係でも、定量評価におけるスコアがより高い後者の手法の方がより薬効分類の名称から推測できる原疾患に近いものが得られました。特にWord2Vecで学習した後近傍をとる場合、正規化を行わない内積では関連度が低く出現頻度の高いアイテムが、正規化を行った場合は類似度がたまたま高いマイナーなアイテムが出てしまうといった問題が起こるのですが、metric learningを用い学習中にコサイン類似度をもって学習を行った場合は、その医薬品に関連のある原疾患に加え、「発熱」・「高血圧」といったコーパス全体での出現頻度が高い原疾患も適切に上位に類似アイテムとして表示されました。
医薬品-有害事象の類似度の確認
医薬品と有害事象のベクトルについてコサイン類似度で近傍を取得しました。
gensim 副作用+原疾患あり
| 0 | 1 | 2 | 3 | 4 | |
|---|---|---|---|---|---|
| アセトアミノフェン | 過敏症 | 患者による企図的医療機器除去 | 乏渇感症 | 多形紅斑 | 巨核球異常 |
| アムロジピンベシル酸塩 | スプルー様腸疾患 | 視床出血 | 一過性脳虚血発作 | 脾臓スキャン異常 | 脳幹出血 |
| アロプリノール | 高血圧 | 中心性チアノーゼ | 高カリウム血症 | 腎硬化症 | CD4リンパ球増加 |
| フルオロウラシル | 2型過敏症 | 注射に伴う反応 | 子宮頚部癌第4期 | 好中球減少性感染 | 遠隔転移を伴う肝癌 |
| 五苓散 | 偽アルドステロン症 | 閉経期症状 | 中毒性皮疹 | 腸間膜静脈硬化症 | 肝脾膿瘍 |
| リバビリン | 肝炎ウィルス関連腎症 | 抗インターフェロン抗体陽性 | プロトロンビン量異常 | C型肝炎RNA増加 | 肝臓血管腫 |
| イオパミドール | ヒスタミン濃度増加 | トリプターゼ増加 | 立毛 | 橈骨動脈脈拍異常 | 血管拡張術 |
| オキシコドン塩酸塩水和物 | 癌疼痛 | 腫瘍熱 | 免疫性腸炎 | 肝転移 | 中枢神経系転移 |
nadare baseline
| 0 | 1 | 2 | 3 | 4 | |
|---|---|---|---|---|---|
| アセトアミノフェン | 中毒性表皮壊死融解症 | スティーヴンス・ジョンソン症候群 | 胆管消失症候群 | 尿細管間質性腎炎 | 皮膚粘膜眼症候群 |
| アムロジピンベシル酸塩 | 血管内ガス | 肉芽腫性皮膚炎 | 毛孔性紅色粃糠疹 | コントロール不良の血圧 | スプルー様腸疾患 |
| アロプリノール | 線維性心内膜炎 | 痛風 | 好酸球増加と全身症状を伴う薬物反応 | 低亜鉛血症 | 痛風性関節炎 |
| フルオロウラシル | 白血球減少症 | 好中球減少症 | 咽頭知覚不全 | 埋込み部位離開 | 閉塞性瘢痕ヘルニア |
| 五苓散 | 高血圧切迫症 | 多形日光疹 | 偽アルドステロン症 | 腸間膜静脈硬化症 | 薬物性肝障害 |
| リバビリン | 網膜症 | 陰嚢出血 | B細胞性前リンパ球性白血病 | パートナーの自然流産 | 抑うつ症状 |
| イオパミドール | 造影剤アレルギー | ショック | アナフィラキシーショック | アナフィラキシー様ショック | くしゃみ |
| オキシコドン塩酸塩水和物 | 丘疹状蕁麻疹 | 溢流性下痢 | 譫妄 | 逆説疼痛 | 癌疼痛 |
考察
医薬品-有害事象間でも後者の手法の方がより添付文章等で確認できる有害事象が現れていると考えられます。一方で、前者のオキシコドン塩酸塩水和物に対する癌疼痛や後者のアロプリノールに対する痛風は有害事象というよりは原疾患に見えます。これらの例についてはデータセット由来の問題か手法の問題化は検討する必要があります。
類似ベクトルの確認のまとめ
薬効分類を用いて定量評価しスコアの高かった手法は、医薬品-医薬品の類似度に加え医薬品-原疾患や医薬品-有害事象間でも良い近傍を得られることが分かりました。この結果を踏まえ、最も評価の高かったnadare baselineについてEmbedding projectorによる可視化する方法を紹介します。
Embedding projectorによる可視化
Embedding projectorはgoogleの開発するTensorBoardの機能の1つのEmbedding projectorのブラウザ版で、ベクトルとベクトルに関するメタデータを読み込むことでインタラクティブにベクトルの可視化を行うことができます。
今回はnadare baselineで学習させたベクトルを公開します。
次のリンクからEmbedding projectorのページに飛んでください。リンク
なお、これから下の図表は公開しているベクトルよりも古いバージョンでの結果のため、上記のリンクで得られる結果とは異なる場合があります。
読み込みに成功すると以下のようにベクトルの散布図が表示され、点にマウスオーバーするとそのベクトルの値が表示されます。
そのままベクトルをクリックするとそのベクトルの近傍のベクトルが表示されます。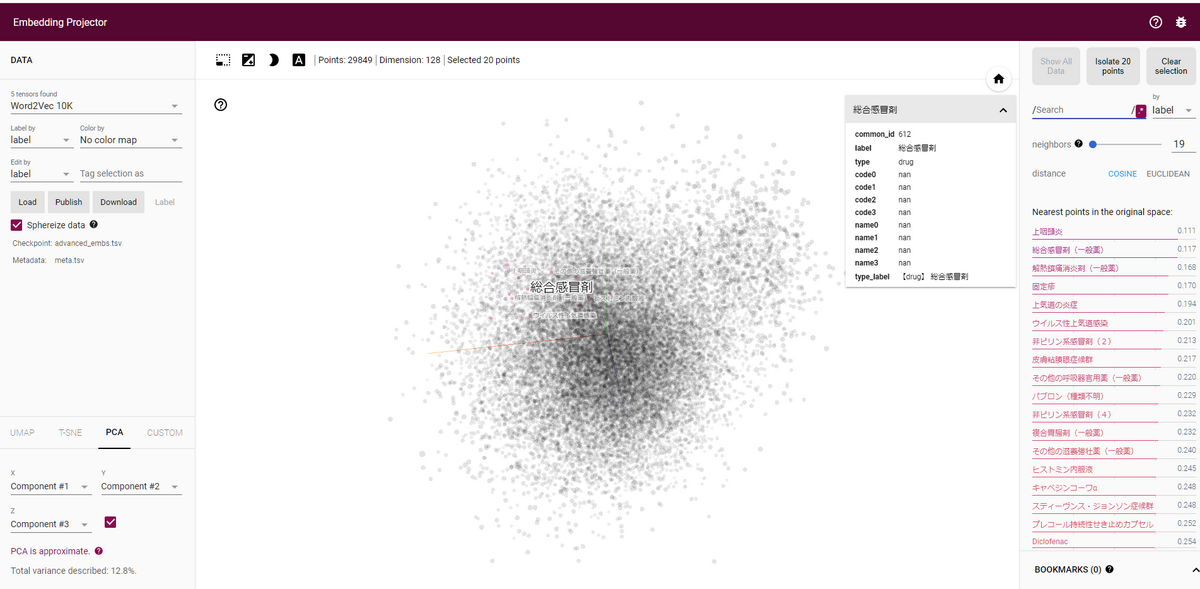
メタデータから検索する。
今回はメタデータとして以下のデータを用意しました。
| 列名 | 内容 | |
|---|---|---|
| 0 | common_id | drug-reac-hist全体でユニークなID、重複あり |
| 1 | label | 医薬品の一般名、原疾患名、有害事象の名前 |
| 2 | type | drug, reac, histのいずれか |
| 3 | code0 | 対応する薬効分類番号の上1桁の数字, 一部の医薬品のみ |
| 4 | code1 | 対応する薬効分類番号の上2桁の数字, 一部の医薬品のみ |
| 5 | code2 | 対応する薬効分類番号の上3桁の数字, 一部の医薬品のみ |
| 6 | code3 | 対応する薬効分類番号の上4桁の数字, 一部の医薬品のみ |
| 7 | name0 | 対応する薬効分類番号の上1桁の名称, 一部の医薬品のみ |
| 8 | name1 | 対応する薬効分類番号の上2桁の名称, 一部の医薬品のみ |
| 9 | name2 | 対応する薬効分類番号の上3桁の名称, 一部の医薬品のみ |
| 10 | name3 | 対応する薬効分類番号の上4桁の名称, 一部の医薬品のみ |
| 11 | type_label | type と labelを同時に表記 |
画面左のLabel byで表示名を変更できるので、type_labelに設定します。
その上で、画面右の検索boxから医薬品を検索してみましょう。
筆者は腰痛持ちなので、たびたびお世話になる「サリチル酸メチル」を入力します。
候補がいくつか出てくるので、その中からサリチル酸メチルを選択するとサリチル酸メチルの近傍の値が表示されます。バップ剤(湿布の一種)や湿布に関連した成分が出ています。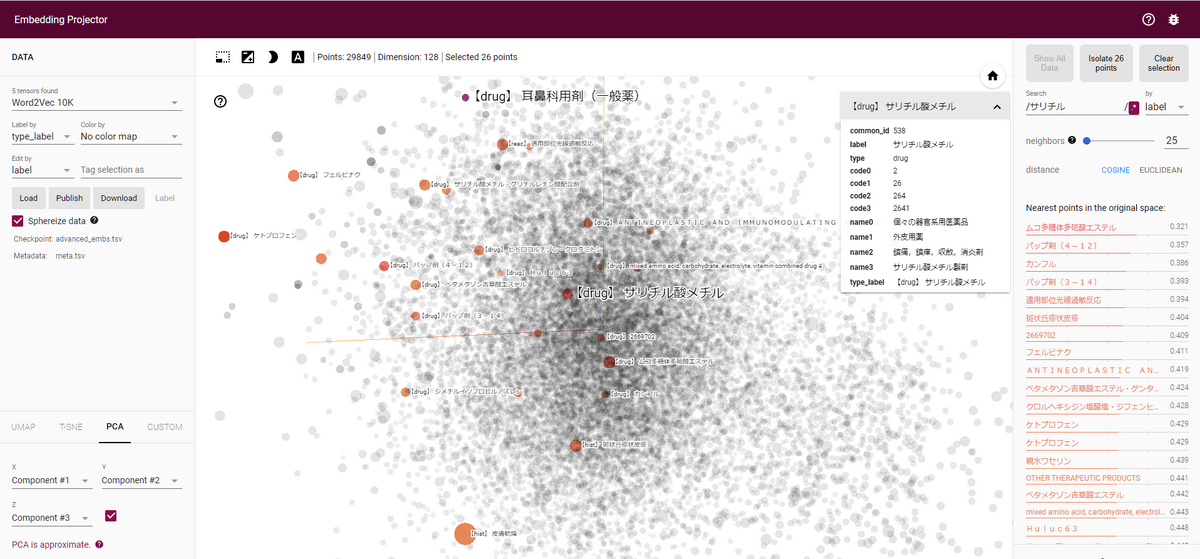
右のneighborのスライダーを調節した上で「Isolate X points」をクリックすると近傍のみ表示できます。
メタデータから絞り込み・色分けする
薬効分類番号のあるデータに絞って表示し、分類番号ごとに色分けして上手く表示できるか確認します。
右のSearchの正規表現オンオフボタン「.*」を押し、正規表現での検索を有効にします。
Searchに「^[0-9]」を入力すると薬効分類番号のあるデータのみが選べるので、その状態で「Isolate 2230 points」をクリックすると絞り込みができます。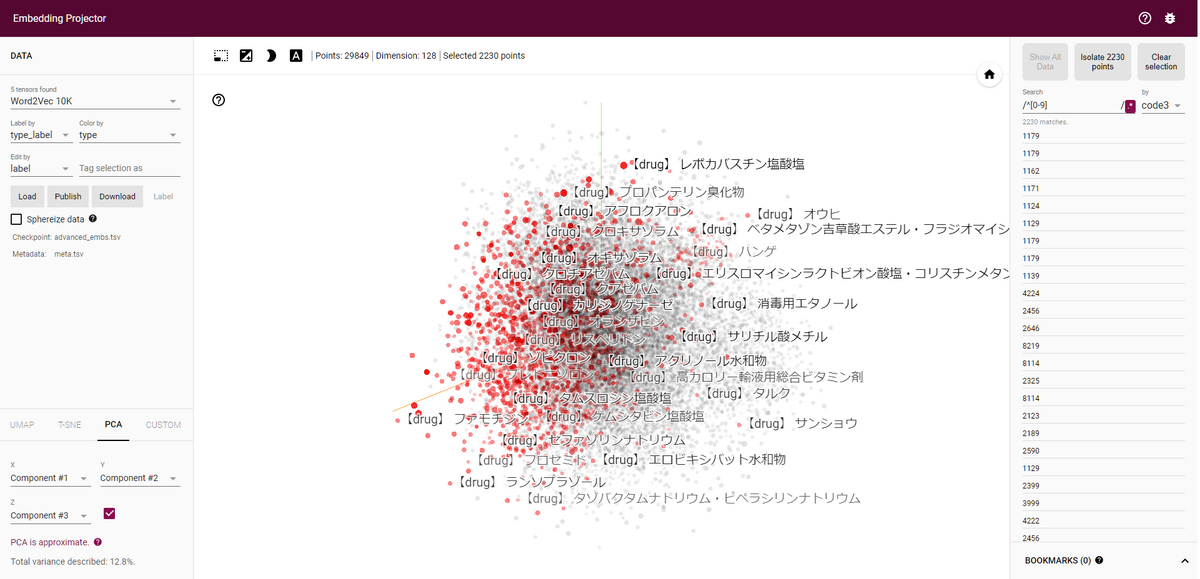
絞り込んだ状態で色を付けて確認してみましょう。
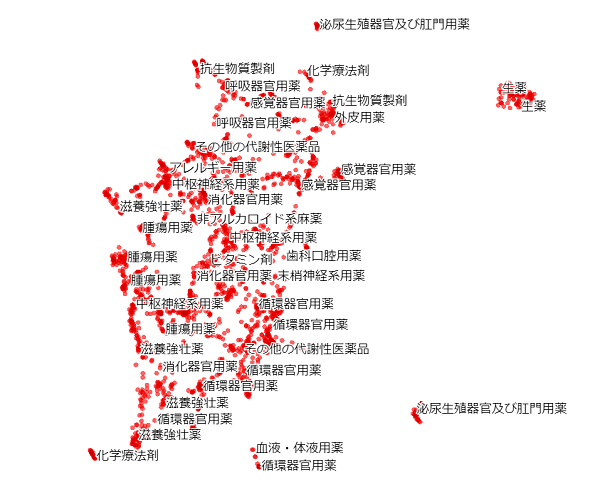
左のLabel byを「name1」にし、Color byを「code1」にすると薬効分類番号の上二桁で色付けされた状態で表示されます。
左下のチェックボックスを外すと2次元で描画できるので、その状態の図を表示します。
左下のUMAP, T-SNEを押すと他の次元圧縮の方法が使えます。
UMAPを押すと次元圧縮の計算が行われます。
Neighborsを50で計算すると下の図のように見やすくなりました。UMAPやt-SNEでの圧縮後の距離は元空間の距離から崩れてしまうので注意が必要ですが、これによって得られたベクトルが他のタスクに使えるかなどをざっくり判断できます。

おわりに
今回はword2vecを用いて医薬品副作用データベースから医薬品の埋め込み表現を獲得できました。さらにtensorflowで様々なテクニックを使うことでより良い表現を得られ、その効果について定量・定性評価により確認できました。
ただ、gensim実装に比べてtensorflow実装は計算時間が大幅にかかり、現状だとtensorflowの方はGPUを使ってもgensimの30~60倍の計算時間がかかります。今後はこの辺りを改善しつつ、より良い埋め込み表現の獲得を目指していきたいと思います。
- 1.Tomas Mikolov, Ilya Sutskever, Kai Chen, Gregory S. Corrado, Jeffrey Dean. Distributed Representations of Words and Phrases and their Compositionality. NIPS, 2013 ↩
- 2.Tomas Mikolov, Ilya Sutskever, Kai Chen, Gregory S. Corrado, Jeffrey Dean. Efficient Estimation of Word Representations in Vector Space. CoRR, 2013 ↩